在作物研究、微生物学、药物开发等场景的科研探索中,科研人员常常要解析DNA–蛋白质的互作,以揭示基因调控网络和表观遗传机制。然而,传统DNA–蛋白质互作技术总有“难以避免”的痛点:
1、ChIP-seq:耗时长,需要大量细胞样本,且背景噪声大,信噪比低,数据解析复杂。
2、传统CUT&RUN:虽然灵敏度有所提升,但操作仍繁琐,流程复杂,重复性和效率受限。
为了解决这些问题,近岸蛋白潜心研发出NEXT CUT&RUN(NovoNGS® NEXT CUT&RUN High-Sensitivity Kit (for Illumina®),Cat.No.: N359)——新一代精简型CUT&RUN方案,让DNA–蛋白互作分析更高效、更精准、更轻松。
五大产品优势,专为高效科研设计
1、相比传统CUT&RUN技术,背景更低,信噪比大幅提升;
2、低至5000个细胞,操作简单,仅需10 h;
3、融合pAG-Nuclease,一步实现靶标识别和高效切割;
4、抗体单次实验使用量低至0.1-0.5 μg,节省成本;
5、随附建库试剂,从靶标捕获到文库构建一套搞定。
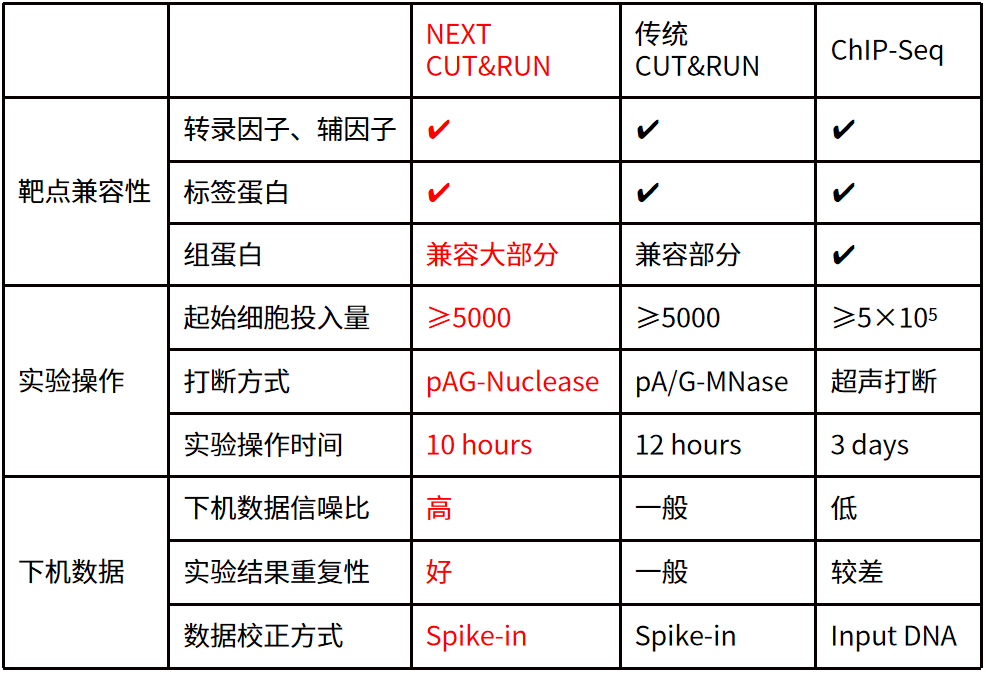
DNA/蛋白互作研究技术比对
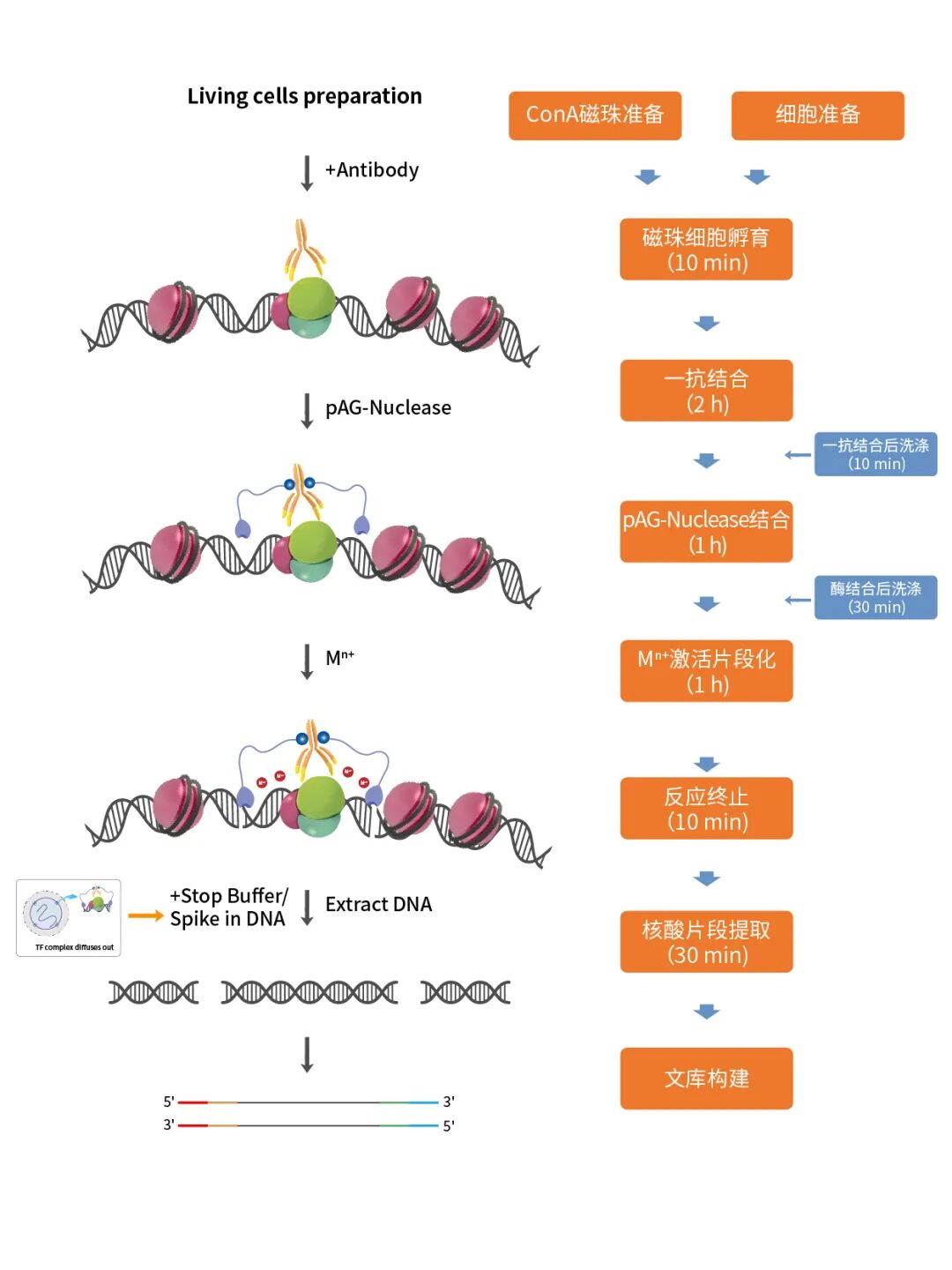
实验流程图
性能展示
1 NEXT CUT&RUN文库构建质量高
转录因子研究:
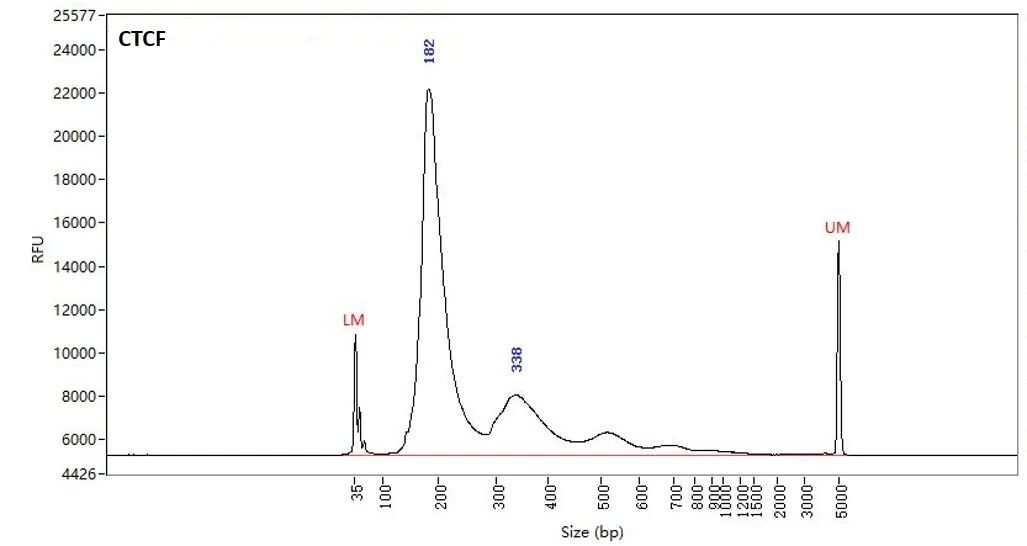
图1. NEXT CUT&RUN试剂盒在100K K562细胞条件下进行CTCF CUT&RUN建库。毛细管电泳结果显示,文库呈现典型的核小体周期分布,主峰180 bp以上,符合预期片段大小,表明文库构建成功且切割效率好,可用于后续高质量测序分析。
组蛋白研究:
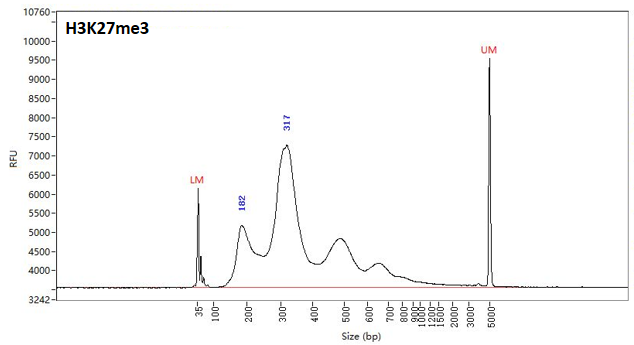
图2. NEXT CUT&RUN试剂盒在100K K562细胞条件下进行H3K27me3 CUT&RUN建库。文库呈现典型的核小体周期分布,主峰约320 bp,符合预期片段大小,表明文库构建成功且切割效率好,可用于后续高质量测序分析。
2 下机数据背景低,信噪比高
IGV数据分析:
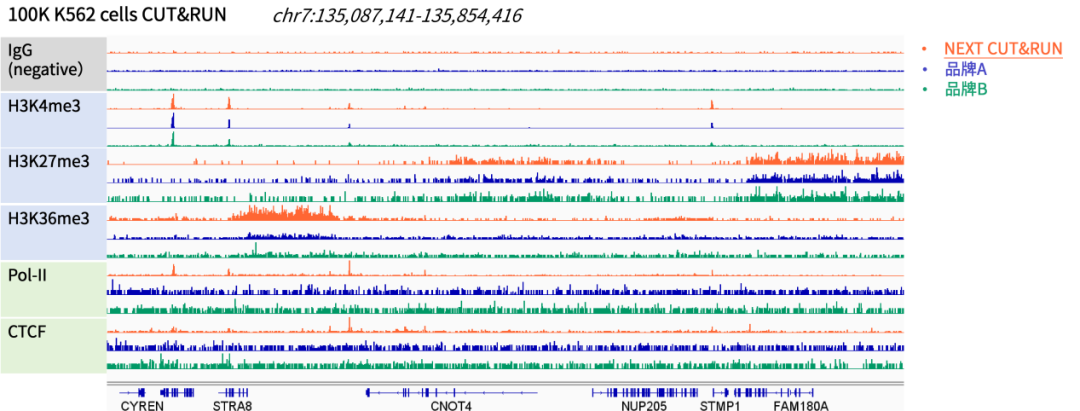
图3. 以100K个K562细胞为样本,使用不同品牌的CUT&RUN试剂盒对组蛋白修饰、DNA结合蛋白及转录因子进行检测。IGV结果显示,使用近岸蛋白NEXT CUT&RUN可在不同组蛋白、DNA结合蛋白和转录因子中获得背景清晰,信噪比高的互作图谱,且信噪比明显优于其他同类产品。
TSS富集分析:
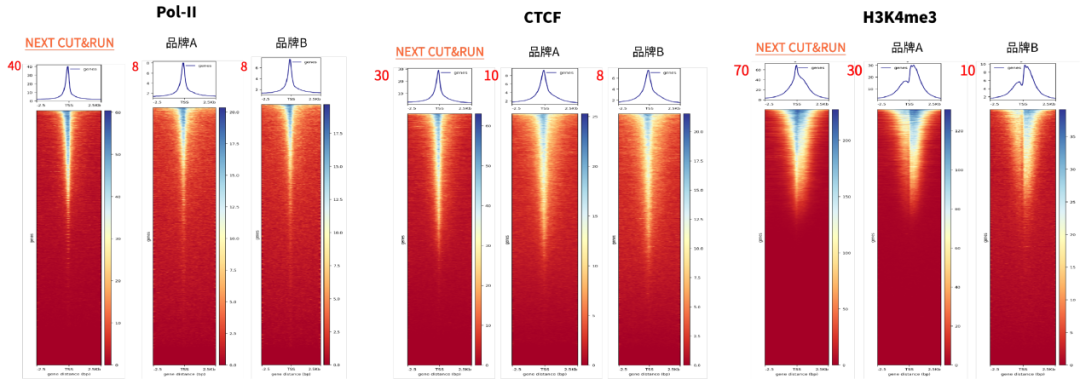
图4. 以30K个K562细胞为样本,使用不同品牌的CUT&RUN试剂盒对组蛋白修饰、DNA结合蛋白及转录因子进行检测。通过TSS富集分析发现,近岸蛋白NEXT CUT&RUN试剂盒获得的TSS富集得分显著高于其他同类产品,表现出更优的信噪比。
3 NEXT CUT&RUN建库所需样本量少,仅需5000个细胞
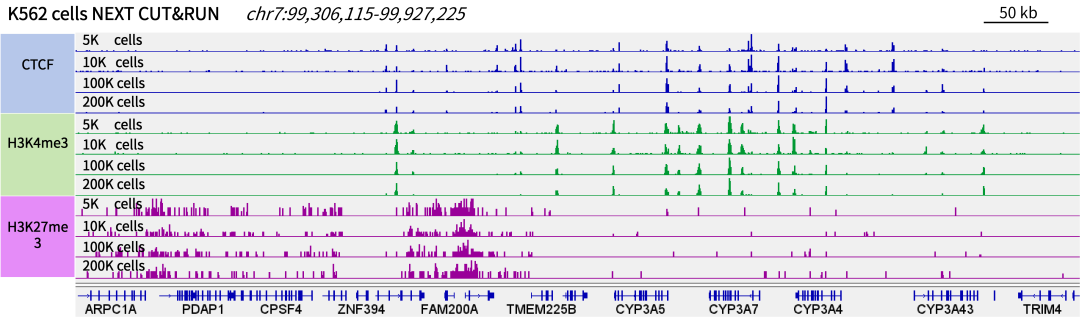
图5. 以K562细胞为样本,使用NEXT CUT&RUN在不同细胞数量条件下对组蛋白修饰及转录因子进行检测。IGV结果显示,仅需5000个细胞量也可获得清晰、高质量结果,且峰型与高细胞量条件保持一致。
4 NEXT CUT&RUN兼容不同类型细胞的互作研究
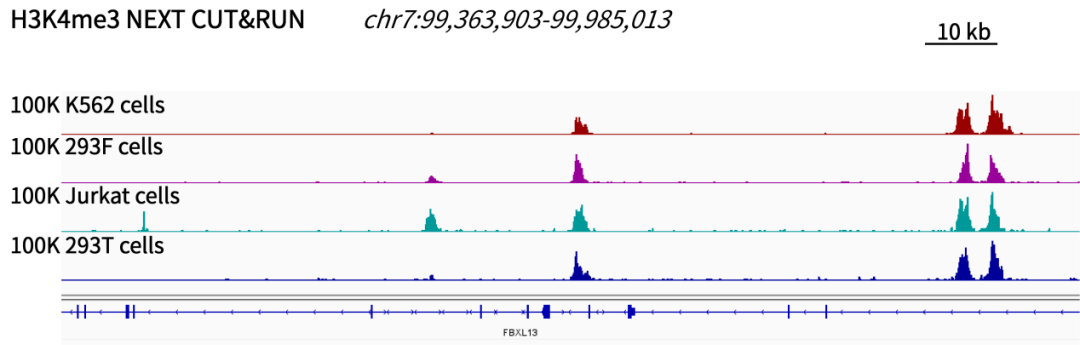
图6. 使用NEXT CUT&RUN试剂盒对100K数量不同人源细胞进行H3K4me3抗体检测,IGV结果显示,四种不同来源的细胞均可以获得高质量的互作图谱。
产品信息

如此高质量的NEXT CUT&RUN试剂盒,你是否心动了?可以扫描下方二维码申请样品哦~数量有限,先到先得!

